| Klick mich an! |
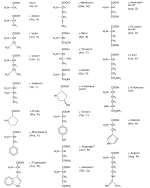 |
| Abb. 10: Die wichtigsten Aminosäuren und ihre Abkürzungen |
| Klick mich an! |
 |
| Abb. 10: Die wichtigsten Aminosäuren und ihre Abkürzungen |
3.3 Aminosäuren - Peptide - Proteine
3.3.1 Aminosäuren und deren Polykondensate
Aminosäuren sind bifunktionelle Verbindungen. Sie tragen zugleich je eine saure und eine basische Gruppe. Das Bauschema der Aminosäuren ist stets das gleiche:
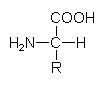
Es gibt 21 verschiedene Aminosäuren, aus denen die Peptide bzw. Proteine aufgebaut sind (-> Abb. 10). Man unterscheidet je nach Fragestellung polare und unpolare, aliphatische und aromatische, saure und basische, negativ oder positiv geladene, alkoholische, amidische, heterocyclische oder schwefelhaltige Aminosäuren. Acht von ihnen sind essentiell: Valin, Leucin, Isoleucin, Methionin, Phenylalanin, Threonin, Lysin, Tryptophan. (Lysin wird z. B. dem Mehl nach dem Mahlvorgang zugesetzt.)
Aminosäuren werden unter bestimmten Bedingungen durch Reaktion (Kondensation) zwischen Amino- und Säuregruppe verbunden (Amidbindung). Hierzu ist die Aktivierung der COOH-Gruppe notwendig. Dies geschieht z. B. durch Säurechloridbildung (R-COCl) oder (biochemisch) durch Bindung an die Transfer-RNA (-> 3.4.2). Peptide und Proteine sind also Polyamide und damit vergleichbar mit Nylon und Perlon.
1. Die Aminosäuren werden aktiviert:
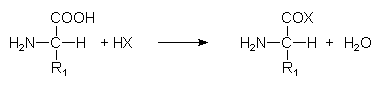
2. Die aktivierten Aminosäuren reagieren zum Peptid:
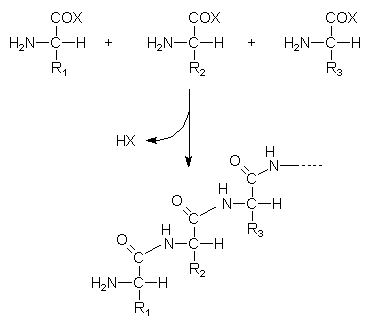
Kleinere Polykondensate nennt man Peptide, die Bindung heißt deswegen auch Peptidbindung. Polypeptide mit Molmassen über 10.000 Daltons nennt man Proteine (Eiweiße). Proteide sind Proteine, die Bestandteile enthalten, die nicht durch Aminosäurekondensation eingeführt sind. Beispiele sind Hämoglobin mit dem eisenhaltigen Hämkomplex, Enzyme mit Coenzymen auf Vitaminbasis sowie Antikörper mit Kohlenhydratanteil.
Beispiele für natürliche Peptide
Beispiele für Proteine
3.3.2 Struktur der Proteine
Form, Struktur und Funktion der Proteine werden durch die Reste der Aminosäuren sowie deren Wechselwirkungen und damit letztlich durch die Sequenz der Aminosäuren bestimmt, die wiederum durch den auf der DNA festgelegten genetischen Code bei der Proteinbiosynthese gesteuert wird (-> 3.4.2). Die chemisch labilen Aminosäurereste sind die Punkte, an denen Denaturierungsmechanismen und Enzymgifte angreifen.
| Klick mich an! |
 |
| Abb. 11: Die Aminosäuresequenz der A- und B-Kette des Rinderinsulins |
Man unterscheidet bei den Proteinen vier Strukturtypen, die in jedem Molekül vorhanden sind und ihm die spezifische Struktur geben.
Primärstruktur
Die Primärstruktur wird durch die Reihenfolge der Aminosäuren,
also durch den genetischen Code der DNA bestimmt (-> 3.4.2).
Sekundärstruktur
Wasserstoffbrückenbindungen zwischen den Peptidbindungen
bestimmen die Sekundärstruktur. Bilden sich Brücken zwischen
parallel liegenden Peptidketten aus, so formen sich die Faltblattstrukturen.
Diese finden wir in besonders elastischen Proteinen
wie den Spinnweben und der Seide. Werden die Brücken innerhalb
einer Peptidkette gebildet, so erhält man eine Helix, wie sie etwa in
Sehnen vorliegen.
Die meisten Proteine stellen allerdings Mischtypen dar.
Tertiärstrukturen
Tertiärstrukturen werden durch unterschiedlichste Bindungen
zwischen Aminosäureresten bestimmt. Diese geben dem Protein
die endgültige Form (kugelig, ellipsoid, gestreckt). Folgende
Bindungen sind bei Tertiärstrukturen möglich:
Hydrophile Bindungen, d. h. H-Brücken- und Salzbindungen wirken besonders nach außen, während sich die unpolaren van der Waals-Bindungen nach innen absättigen.
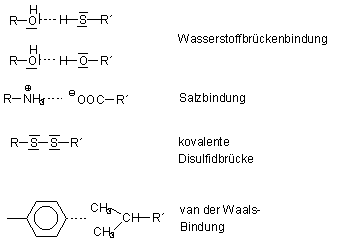
Abb. 14: Ausbildung der Tertiärstrukturen von Proteinen
Quartärstrukturen
Quartäre Strukturen sind mehr oder weniger lockere Verbände aus
gleichen oder verschiedenen Proteinen. Z. B. ist das Hämoglobin Hb
aus vier Ketten aufgebaut, von denen jeweils zwei identisch sind: a-
und b-Ketten.
3.3.3 Denaturierung von Proteinen
Die Funktion eines Proteins hängt stark von der ungestörten Geometrie des Moleküls ab. Andererseits sind Proteine in unserem Verdauungstrakt nur abbaubar, wenn sie zuvor denaturiert wurden (z. B. durch Kochen oder durch die starke Salzsäure in unserem Magen). Diese Denaturierung ist von großer Bedeutung für die Nahrungshygiene, da hierbei auch Bakterien und Pilze zerstört werden.
Denaturierung greift an den verschiedenen Bindungstypen innerhalb der Proteine an. Allerdings ist Denaturierung nicht notwendigerweise mit Ausflockung verbunden. Denaturierte Enzyme z. B. können gelöst bleiben, tragen allerdings in ihrem aktiven Zentrum ein ihre Aktivität einschränkendes Molekül oder Ion. Das Riesenmolekül Urease (Molmasse 100.000) wird bereits durch ein bis zwei Cu2+-Ionen inaktiviert. Ein Phosphorsäureester am Serinrest im aktiven Zentrum inaktiviert die Acetylcholinesterase (-> 7.1.1).
Denaturierungsmechanismen:
Erhitzen
löst das komplizierte intramolekulare Bindungssystem der Proteine
durch Schwingungsanregung. Beim Abkühlen werden neue
Bindungen geknüpft, wobei intermolekulare Zufallsstrukturen
entstehen: Hautbildung auf gekochter Milch, Hartwerden von
gebratenem Fleisch.
Alkohole, inerte Salze
Bei der Einwirkung von Alkoholen und inerten Salzen (wie Natriumchlorid
oder Natriumsulfat) in hohen Konzentrationen werden die
Primär- bis Quartärstrukturen aufgrund der Konkurrenz um
Wasserstoffbrückenbindungen zerstört. Deshalb kann man mit Salz oder
Alkohol proteinhaltige Esswaren konservieren.
Änderung des pH-Wertes
Hierbei wird die Bildung von salzartigen Bindungen zwischen NH3+-
und COO¯-Resten verhindert. So beobachtet man z. B. flockige
Koagulation in saurer Milch.
Fällung durch Schwermetall-Ionen
Hiervon sind vor allem schwefel- und stickstoffhaltige funktionelle
Gruppen betroffen. Es kommt gerade bei mehrwertigen Metall-Ionen
zu Quervernetzungen zwischen verschiedenen Proteinmolekülen
und damit zur Ausflockung.
Fällung durch Tenside
Grenzflächenaktive Substanzen stören die ins Innere der Proteine
gerichteten apolaren Bindungen. Damit werden die Moleküle regelrecht umgekrempelt.
Reaktion mit Sulfiden R-SH
Beispiele sind Schwefelwasserstoff oder Thioglykolsäure. Hierbei
kommt es zur Spaltung von -S-S-Brücken:
R-S-S-R + H2S ———> R-SH + HS-S-R
Die Umsulfidierung kann durch Oxidationsmittel rückgängig gemacht werden. Dies nutzt man bei der Dauerwelle aus.
3.3.4 Enzyme
Enzyme sind katalytisch wirksame Proteine. Sie setzen die Aktivierungsenergie ganz besonders stark herab. Grund ist die optimale sterische Ausrichtung der Edukte vor und während der Reaktion in aktiven Zentren, deren Geometrie von speziellen Resten einiger weniger Aminosäuren und deren Lage zueinander bestimmt wird.
Ein Beispiel ist die in der Leber ablaufende Oxidation von Ethanol zu Acetaldehyd durch das Enzym ADH (Alkoholdehydrogenase). Durch Einlagerung zwischen je einer funktionellen Gruppe mit Elektronenmangel und Elektronenüberschuss wird das Ethanolmolekül extrem polarisiert, so dass es zu einem simultanen "Umklappen" der Bindungen kommt.
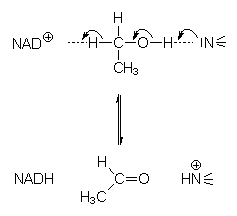
Abb. 15: Umklappen der Bindungen bei der ADH-Reaktion
| Klick mich an! |
 |
| Abb. 16: Reaktionsmechanismen der ADH |
Den genauen Mechanismus zeigt die Abbildung 16. Die ADH bindet nicht nur NAD+ (über ein -S¯ und ein Zink-Ion), sondern es steuert auch die Ausrichtung des Ethanols, indem es dessen lipophile Methylreste bindet. (Da diese beim Methanol fehlen, wird es nicht umgesetzt. Deshalb ist Methanol so giftig.)
Spezifitäten
| - | Enzyme sind milieuspezifisch. Dies betrifft vor allem pH-Wert und Redoxlage der Umgebung, aber auch ihre Fixierung in Membranen oder Multienzymkomplexen. |
| - | Enzyme sind im Allgemeinen substratspezifisch (bei Synthesen oder speziellen Stoffwechselleistungen) oder gruppenspezifisch (z. B. bezogen auf Bindungstyp bei Verdauungsenzymen) (Schlüssel-Schlossprinzip). |
| - | Enzyme sind reaktionsspezifisch. Proteasen spalten nur Proteine, nicht aber Triglyceride. |
Nach der Reaktionsspezifität unterscheidet man folgende Enzymgruppen:
| 1. | Oxidoreduktasen (Dehydrogenasen) | |
| - | oxidieren Substrate mit NAD(P), Cytochromen,
O2, H2O2 etc.
(Beispiele: Alkoholdehydrogenase, Katalase, Peroxidasen). |
|
| 2. | Transferasen | |
| - | transferieren eine Gruppe zwischen einem Substratpaar
(Acetylcholintransferase, Glucosephosphatase). |
|
| 3. | Hydrolasen | |
| - | spalten Bindungen unter Wasseranlagerung
(Lipase, Acetylcholinesterase, Saccharase, Pepsin). |
|
| 4. | Lyasen | |
| - | spalten C-C-, C-O- und C-N-Bindungen unter Ausbildung
von
Doppelbindungen (Decarboxylasen). |
|
| 5. | Isomerasen | |
| - | bilden Isomere
(Racemasen, Epimerasen). |
|
| 6. | Ligasen | |
| - | knüpfen Bindungen, z. B. C-C oder C-N unter
ATP-Verbrauch
(Carboxylasen, Peptidsynthetasen). |
|
Viele Enzyme wirken ohne Cofaktoren. Ein bekanntes Beispiel ist die höchst unempfindliche RNAse, die interzellulär RNA abbaut und damit stoffwechselregulierend wirkt.
Oft reicht das Protein allein nicht aus, um eine bestimmte Wirkung zu erzielen. Das Enzym bedarf dann eines Cofaktors, der oft von Vitaminen oder Spurenelementen abgeleitet ist. So trägt die Alkoholdehydrogenase ein Zink-Ion, an das NAD+ komplex gebunden ist (-> Abb. 16). Die Cytochromoxidase enthält Eisen-Ionen, komplex gebunden in einem Porphyrinring, und zusätzlich Kupfer-Ionen.
Die Glutathionperoxidase, die in den Erythrocyten organische Peroxide R-OOH abbaut, enthält ein Selenatom in ihrem aktiven Zentrum.